Más allá de la secuencia: estrategias analíticas para garantizar la calidad del ARNm terapéutico

Las terapias basadas en ARN mensajero (ARNm) se reconocieron como la solución a muchos problemas de enfermedades no cubiertas durante la pandemia de COVID-19, cuando su aplicación como vacunas profilácticas demostró su superioridad frente a formulaciones tradicionales.
Sin embargo, esta tecnología no nació en 2020, su desarrollo comenzó hace más de dos décadas, con investigaciones iniciales centradas en la inmunoterapia contra el cáncer [1]. Desde entonces, los avances en la síntesis, modificación y transporte del ARNm han transformado su potencial terapéutico, expandiendo su aplicación a patologías infecciosas, oncológicas, raras y metabólicas.
El principio de acción es conceptualmente simple: el ARNm sintetizado se introduce en las células aberrantes, donde los ribosomas de la propia célula traducen la información codificada para producir la proteína terapéutica deseada. No obstante, este enfoque enfrenta importantes desafíos. El ARNm es una molécula inestable, altamente susceptible a la degradación por ribonucleasas, y capaz de inducir respuestas inmunitarias innatas no deseadas. Además, no es capaz de penetrar la membrana plasmática por sí solo. Superar estas limitaciones ha requerido avances tanto en la ingeniería del ARNm durante la síntesis como en el desarrollo de sistemas de liberación eficaces y biocompatibles.
En la síntesis in vitro del ARNm, se introducen modificaciones químicas en las bases nitrogenadas, como por ejemplo, una protección en 5’ y la adición de una cola de poli adeninas en 3’, con el objetivo de mejorar la estabilidad general del ARNm, la eficiencia de traducción y reducir la inmunogenicidad [2]. La incorporación de nucleósidos modificados, como la pseudouridina (ψ), la N6-metiladenosina (m6A) y la 5-metilcitosina (m5C), disminuye el reconocimiento por sensores inmunes intracelulares y aumenta la estabilidad del transcrito [3].
Para garantizar la llegada del ARNm a las células diana y protegerlo frente a la degradación extracelular, se utilizan nanosistemas de encapsulación. Las nanopartículas lipídicas (LNPs) se consolidaron durante la pandemia [4], pero existen alternativas prometedoras como las nanopartículas poliméricas o las vesículas extracelulares. Estas presentan ventajas adicionales en términos de biocompatibilidad, biodegradabilidad y capacidad de adaptación a funcionalidades específicas, por ejemplo, para el transporte dirigido, además de mejorar la estabilidad del producto en condiciones de almacenamiento menos exigentes [5,6].
La complejidad estructural y funcional del ARNm ha impulsado a las agencias regulatorias a establecer un conjunto de Atributos Críticos de Calidad (CQAs), tanto para el principio activo (drug substance) como para el producto final con el nanosistema (drug producto) [7,8]. Estos atributos deben ser monitorizados para garantizar la seguridad, eficacia y consistencia del producto, ya que influyen directamente en su rendimiento biológico y farmacológico. En el caso del ARNm como principio activo, los CQAs más relevantes incluyen la identidad, la pureza e integridad del transcrito, y las impurezas [9].
La identidad hace referencia a la correcta estructura molecular del ARNm, incluyendo la presencia de la protección a 5’, las modificaciones nucleotídicas y la longitud de la cola poli-Adeninas en 3’. Cada uno de estos elementos influye en la estabilidad y en la eficiencia de traducción.
En la pureza e integridad se evalúa que el transcrito sea de longitud completa y que los productos de degradación sean mínimos.
En las impurezas, se controlan contaminantes derivados del proceso de síntesis, como ADN plantilla residual, enzimas de transcripción, ARN de doble cadena (dsRNA), nucleótidos (NTPs) o análogos de la protección libres. Estas pueden reducir la eficacia o inducir respuestas inflamatorias [10].
El control riguroso de estos parámetros es esencial y garantizar los CQAs del ARNm requiere herramientas analíticas de última generación. Entre ellas, las metodologías basadas en cromatografía líquida acoplada a diferentes detectores, desde los más básicos (UV/FLU), hasta la espectrometría de masas (LC–MS), se han convertido en herramientas útiles por su versatilidad, sensibilidad y especificidad, adaptando metodologías desarrolladas para molécula pequeña y ampliamente utilizadas en la industria farmacéutica [11]. Estas estrategias se dividen en tres niveles complementarios: digestión total, digestión parcial y análisis intacto.
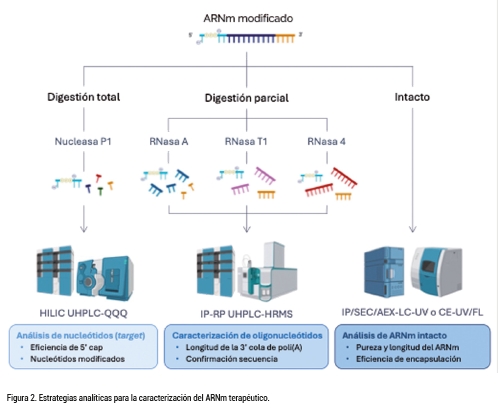
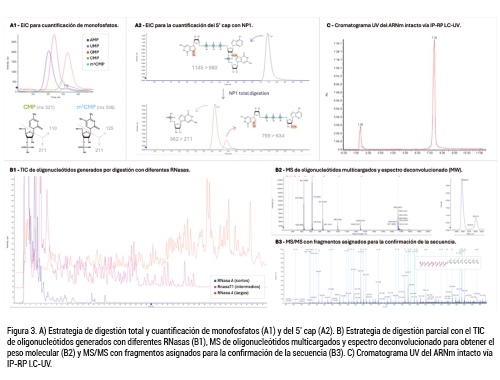
En la digestión total, el ARNm se hidroliza completamente mediante enzimas no específicas, como la nucleasa P1, generando un conjunto de nucleótidos libres. Estos se separan mediante cromatografía con fase estacionaria HILIC-Amide, la cual permite una retención eficaz de estos compuestos altamente polares. De forma alternativa, puede incorporarse una segunda etapa de desfosforilación mediante una fosfatasa, que elimina los grupos fosfato y convierte los nucleótidos en nucleósidos. Este proceso reduce su polaridad y posibilita su separación mediante cromatografía de fase reversa convencional. Los productos obtenidos se detectan posteriormente mediante espectrometría de masas triple cuadrupolo (MRM), lo que permite una cuantificación precisa de los nucleótidos y la identificación de modificaciones específicas, ofreciendo una visión global de la composición del ARNm [12]. No obstante, a pesar de su alta sensibilidad, esta estrategia presenta como principal limitación la pérdida de información sobre la secuencia y el contexto estructural.
La digestión parcial genera fragmentos de oligonucleótidos de longitud definida que conservan información de secuencia y modificaciones. Para ello, se utilizan endonucleasas específicas como RNasa T1 (corta específicamente en guanosinas), RNasa A (corta en ambas pirimidinas, en uracilos y citosinas) o RNasa 4 (corta específicamente en las combinaciones Uracilo con purinas, U-A y U-G). La especificidad del sitio de corte determina directamente la longitud de los fragmentos obtenidos: cuanto más amplia es la especificidad de la enzima, más cortos son los oligonucleótidos generados; por el contrario, una mayor selectividad produce fragmentos más largos, lo que, aunque mejora la información estructural, puede dificultar su ionización y la posterior detección. Los fragmentos resultantes se separan mediante cromatografía líquida en fase reversa con par iónico (IP-RP UHPLC) y se analizan por espectrometría de masas de alta resolución (QTOF) con modos de adquisición dependiente (DDA) e independiente (DIA), para adquirir los espectros de MS que confirman las multicargas de los oligonucleótidos, se deconvoluciona para obtener su masa molecular y los espectros de MSMS para confirmar la secuencia [13]. Gracias a esta combinación de RNasas, se consigue una cobertura de secuencia amplia y una identificación fiable de las modificaciones y su posición, generando este “fingerprint” característico, consolidándose como estrategia para el oligomapping del ARNm terapéutico.
El análisis del ARNm intacto se realiza mediante técnicas convencionales de separación, como la cromatografía líquida o la electroforesis capilar, con detección por absorción ultravioleta (UV) o fluorescencia (FLU). Existen diversas estrategias cromatográficas aplicables a este tipo de biomoléculas, entre ellas la cromatografía de fase reversa con par iónico (IP-RP), que emplea columnas de gran tamaño de poro (>1000 Å) para facilitar la elución de macromoléculas, así como la cromatografía de exclusión por tamaño (SEC) y la cromatografía de intercambio aniónico (AEX). Estas metodologías permiten evaluar la longitud, integridad y contenido global del ARNm intacto [14], y se han demostrado especialmente útiles para cuantificar el ARNm encapsulado en nanopartículas, como producto final (drug product).
La combinación de las tres estrategias analíticas ofrece una caracterización integral del ARNm terapéutico. Esta complementariedad no solo resulta esencial para cumplir con los requisitos regulatorios de caracterización y control de calidad, sino que también refleja la versatilidad de las metodologías analíticas actuales para abordar la caracterización de estas biomoléculas de naturaleza tan compleja.
Persisten, sin embargo, desafíos significativos, especialmente en la interpretación de espectros de fragmentación, que requiere herramientas bioinformáticas avanzadas capaces de manejar grandes volúmenes de datos y minimizar falsos positivos. Estas limitaciones han impulsado notables avances en los últimos 5 años, en estas estrategias híbridas, que amplían los límites de la caracterización de oligonucleótidos y consecuentemente del ARNm.
En el futuro, la estandarización, la automatización y el uso de herramientas bioinformáticas serán claves para fortalecer el control de calidad y transformar estos retos en oportunidades para la medicina de precisión [15].
Bibliografía:
- [1] Cannon G, et. al. DNA Cell Biol. 2002. doi:10.1089/104454902762053882.
- [2] Karikó K, et. al. Curr Opin Drug Discov Devel. 2007.
- [3] Pardi N, et. al. Nat Rev Drug Discov. 2018. doi:10.1038/nrd.2017.243.
- [4] Hou X, et. al. Nat Rev Mater. 2021. doi:10.1038/s41578-021-00358-0.
- [5] Navalón-López M, et. al. Nanoscale Adv. 2023. doi:10.1039/D2NA00800A.
- [6] Giacobino C, et. al. Cancers (Basel). 2021. doi:10.3390/cancers13092280.
- [7] European Medicines Agency (EMA). Guidelines on mRNA-based products. 2022.
- [8] United States Pharmacopeia (USP). Analytical procedures for mRNA vaccines. 2024.
- [9] Whitley J, et. al. Transl Res. 2022. doi:10.1016/j.trsl.2021.11.009.
- [10] Pepini T, et. al. J Immunol. 2017. doi:10.4049/jimmunol.1601877.
- [11] Webb ALJ, et. al. J Chromatogr A. 2025. doi:10.1016/j.chroma.2025.465724.
- [12] Marchante-Gayón JM, et. al. Mass Spectrom Rev. 2023. doi:10.1002/mas.21865.
- [13] Gau BC, et. al. Sci Rep. 2023. doi:10.1038/s41598-023-36193-2.
- [14] Currie J, et. al. ChemRxiv. 2023. doi:10.26434/chemrxiv-2021-mgx9q-v2.
- [15] Magoola M, et. al. Cancers. 2025. doi:10.3390/cancers17111882.
| Nombre | Mar Losada, Gemma Gotor y Cristina Fornaguera |
|---|---|
| Empresa | Institut Químic de Sarrià (IQS), Universitat Ramon Llull (URL). |
| Cargo | Grup d'Enginyeria de Materials (Gemat) |
Si continúas navegando, aceptas su uso.
Más información
Política de privacidad | Cookies | Aviso legal | Información adicional| miembros de CEDRO









































